Projets réalisés par la plateforme
en collaboration avec un ou plusieurs laboratoires de la FR Bioenvis
Amphibgenfam
Développement d'un pipeline d'assemblage de novo et d'annotation de données RNA-seq (projet FR Bioenvis, 2017).
Dans le cadre d'une collaboration avec le LEHNA (T. Lefebure) et le LBBE (P. Veber, S. Penel) et en réponse à un AO FR Bioenvis, le PRABI-AMSB (D. Guyot, V. Navratil) a participé au développement d'un pipeline bioinformatique d'assemblage et d'annotation de novo de données RNA-seq. Ce pipeline est implémenté à l'aide de Bistro et a été appliqué à plusieurs espèces non-modèles d'amphibiens étudiés par le LEHNA, incluant Buffo Buffo, Proteus anguinus, Rana dalmatina. Il comprend l'assemblage de novo des lecture courtes Illumina à l'aide Trinity, l'annotation fonctionnelle des transcrits et le clustering sous forme de famille de gènes des protéines prédites. Un service web trinotate a été mise en place sur nos serveurs afin de faciliter l'accès aux annotations (Buffo buffo, Proteus anguinus, Rana dalmatina). Une formation à l'utilisation des différentes étapes du pipeline a été organisée par la plateforme dans le cadre du plan de formation continue de l'Université de Lyon.
Transcriptome-wide deregulation of gene expression by artificial light at night in tadpoles of common toads. Touzot M., Lefebure T., Lengagne T., Secondi J., Dumet A., Konecny-Dupre L., Veber P., Navratil V., Duchamp C. and Mondy N. Sci Total Environ. 2021
SEX-DETector++
Développement de SEX-DETector sous C++ (2018-2020).
Dans le cadre d'une collaboration avec le LBBE (G. Marais,Hélène Badouin, Jos Käfer, François Gindraud), le PRABI-AMSB (D. Guyot) a apporté un support technique au développement C++ de l'application SEX-DETector++.
The wild grape genome sequence provides insights into the transition from dioecy to hermaphroditism during grape domestication. Badouin H., Velt A., Gindraud F., Flutre T., Dumas V., Vautrin S., Marande W., Corbi J., Sallet E., Ganofsky J., Santoni S., Guyot D., Ricciardelli E., Jepsen K., Käfer J., Berges H., Duchên E., Picard F., Hugueney P., Tavares R., Bacilieri R., Rustenholz C. & Marais GAB. Genome Biol 21, 223 (2020).
FluHit
Etude des perturbations transcriptomiques induites par le virus Influenza A (IAV) par une approche Dual-RNAseq (2017-2020).
En collaboration avec le LBBE (V. Lacroix, C. Benoit-Pilven) et l'Institut Pasteur (N. Naffakh), le PRABI-AMSB (V. Navratil) a participé à l'analyse bioinformatique des effets de l'infection par IAV sur le transcriptome humain. La plateforme a contribué à la conceptions du projet, à la planification expérimentale, à l'analyse multivariée des données dual RNA-seq (étude du Read-Through induit par l'infection et des modifications de l'épissage à l'aide de Kissplice et KissDE) ainsi qu'à la mise à disposition des ressources de calcul et de stockage. Dans une perspective de science reproductible les analyses ont été mises à disposition au travers d'un webservice shiny (shiny-service). Les données sont accessible en opendata sur SRA (access).
Influenza virus infection induces widespread alterations of host cell splicing.Ashraf U.*, Benoit-Pilven C.*, Navratil V., Fournier G., Munier S., Sismeiro O., Coppée JY, Lacroix V., Naffakh N. NAR Genomics and Bioinformatics. 2020Advances in Analyzing Virus-Induced Alterations of Host Cell Splicing. Ashraf U., Benoit-Pilven C.,Lacroix V., Navratil V., Naffakh N. Trends Microbiol. 2019
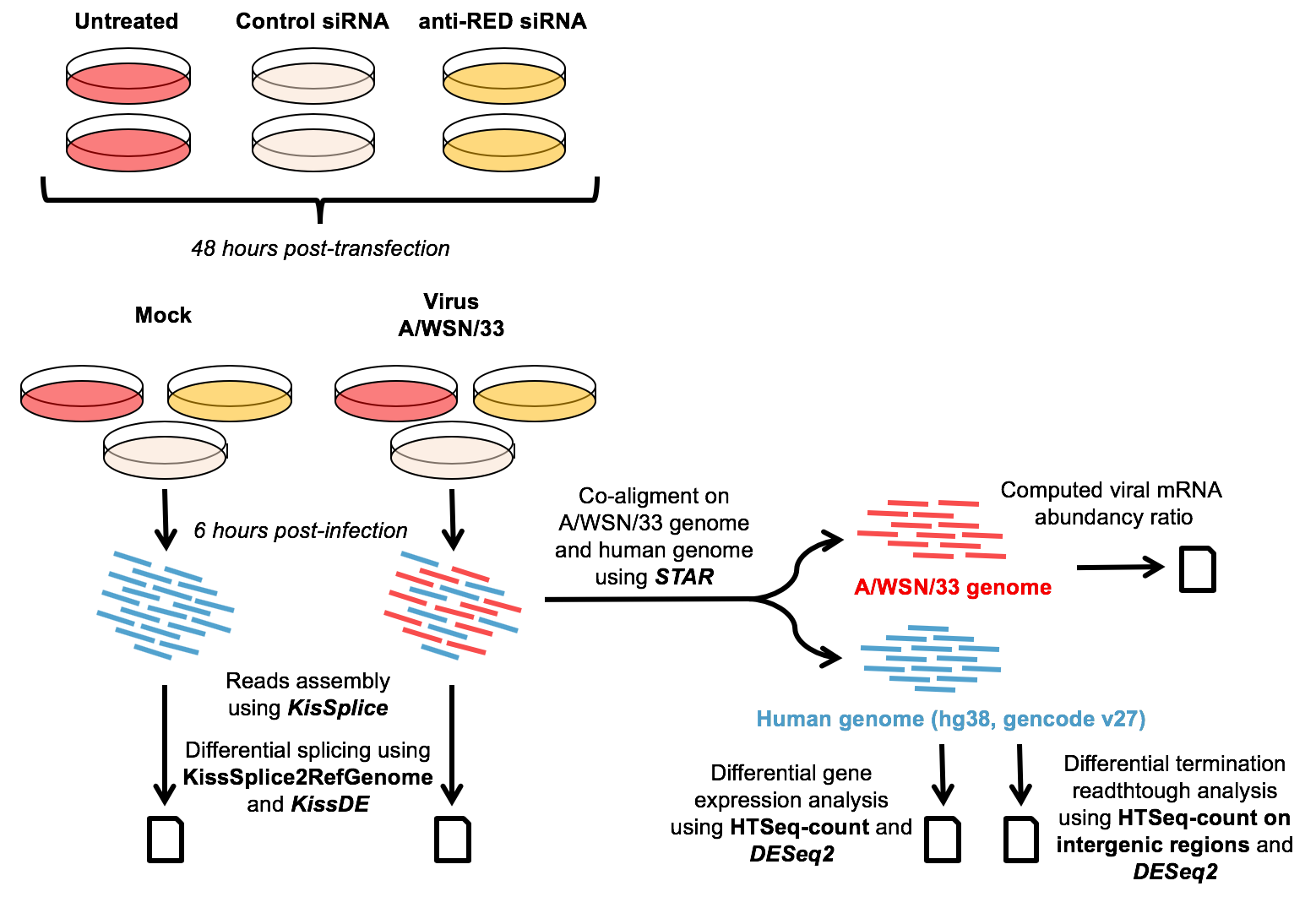
Vibra-FLu
Interactions virome-bactériome-transcriptome impliquées dans la pathogénèse de la grippe chez les enfants par une approche Dual RNA-seq/metagenomique (projet ecofect, 2017-2019).
En collaboration avec le laboratoire VirPath (Bruno Lina, Laurence Josset) et le LBBE (Claire Bardel, Jean Thioulouse, Anne-Béatrice Dufour), le PRABI-AMSB (V. Navratil) a participé au développement bioinformatique d'un pipeline permettant d'étudier le virome et le transcriptome à partir d'échantillons de jeunes enfants atteints de maladies respiratoires aigues. Une base de donnée de génomes viraux a été développée (Viromedb, communication personnelle) et un pipeline utilisant les technologies kraken2/bracken pour l'estimation de l'abundance a été développée (VirHostSeq, communication personnelle). Le PRABI-AMSB a par ailleurs mise à disposition ses ressources de calcul et de stockage. Dans le cadre de ce projet, le PRABI-AMSB participe au comité de pilotage de la thèse de Marina Sabatier (E2M2).
En soutient à la recherche contre la COVID-19, ce pipeline a été appliqué à une cohorte de patients hospitalisés atteints de la maladie afin d'étudier les modifications cellulaires induites par SARS-Cov-2.
Legio
Analyse RNA-seq à grande échelle de souches Legionella pneumophila en croissance intra-macrophagique. (projet FRM, 2014-2017)
En collaboration avec le Centre National de Référence des légionelles (Sophie Jarraud) et le LBBE (Guy Perriere), le PRABI-AMSB (C. Oger, V. Navratil, CDD bioinformatique - A. Fournier). Le PRABI-AMSB a participé à l'analyse bioinformatique des déterminants génétiques associés à la pathogénécité de 32 souches de légionnelles par une approche de DNA-seq et de Dual RNA-seq.
Mosqui-target
Support bioinformatique galaxy pour l'analyse ciblée des SNPs associés à la resistance métabolique des vecteurs de la Dengue. (projet CNRS/INEE-APEGE, 2013-2014)
Collaboration avec le LECA (J.P. David) et le PRABI-AMSB (V. Navratil).
In the hunt for genomic markers of metabolic resistance to pyrethroids in the mosquito Aedes aegypti: An integrated next-generation sequencing approach. Faucon F, Gaude T, Dusfour I, Navratil V., Corbel V, Juntarajumnong W, Girod R, Poupardin R, Boyer F, Reynaud S, David JP. PLoS Negl Trop Dis. 2017Identifying genomic changes associated with insecticide resistance in the dengue mosquito Aedes aegypti by deep targeted sequencing. Faucon F., Dusfour I., Gaude T., Navratil V., Boyer F., Chandre F., Sirisopa P., Thanispong K., Juntarajumnong W., Poupardin R., Chareonviriyaphap T., Girod R. , Corbel V., Reynaud S., David JP. Genome Research 2015

Nucleolin
Etudes des fonctions non-conventionnelles de la Nucléoline (projet FINOVI-AO#08, R12128CC, 2012-2015)
En collaboration avec le CIRI (Anna Greco) et l'ENS Lyon (P. Bouvet), le PRABI-AMSB (V. Navratil) a participé à la recherche de fonctions non-conventionnelles de la nucléoline dans des cellules infectées par HSV-1 en utilisant une approche de biologie systémique basée sur la modélisation du réseau d'interaction protéine-protéine HSV-1/NUCL.
Nuclear Functions of Nucleolin through Global Proteomics and Interactomic Approaches. Salvetti A. , Couté Y., Epstein A., Arata L., Kraut A., Navratil V., Bouvet P., Greco A. J Proteome Res. 2016;15(5):1659-1669.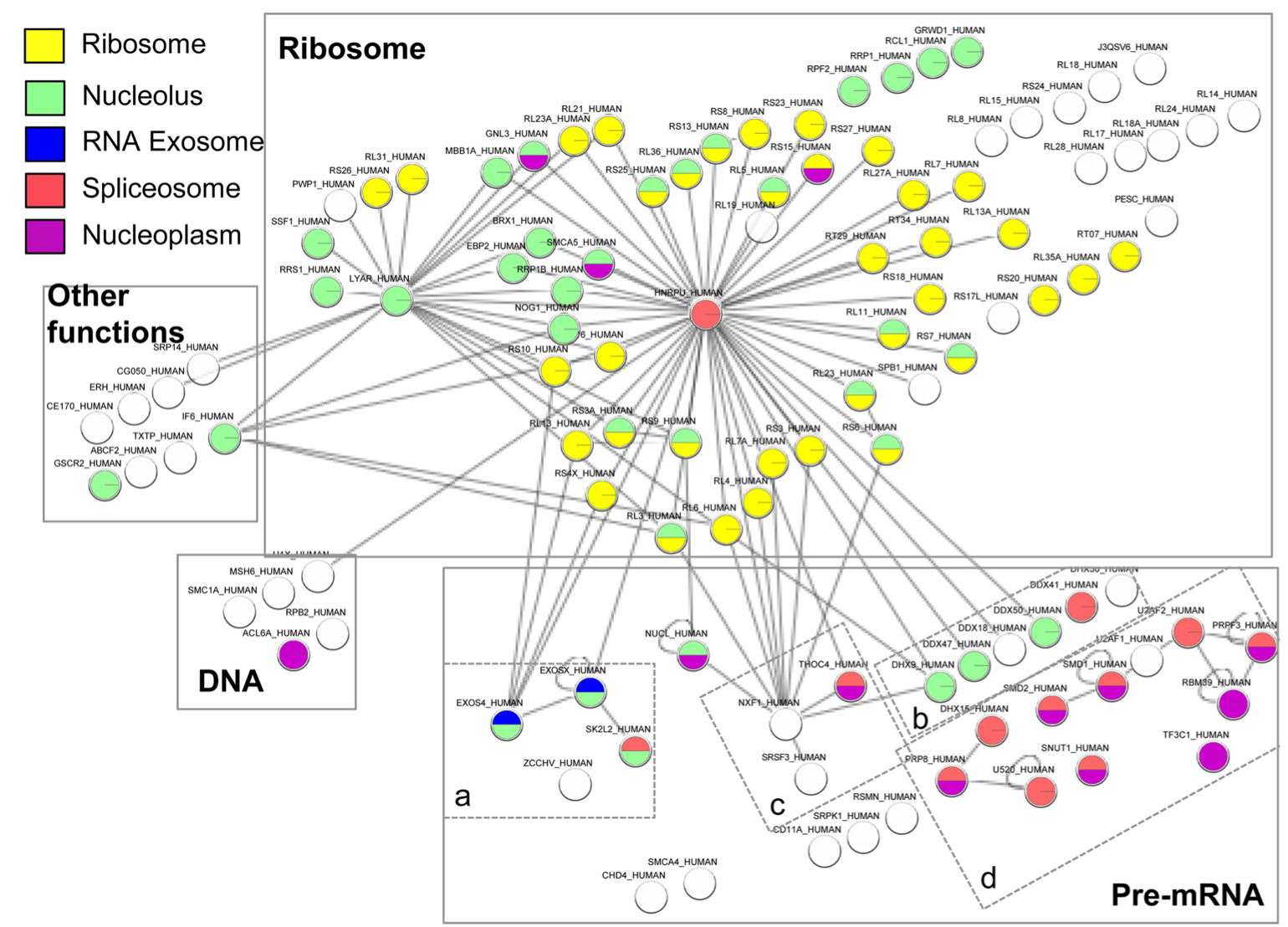
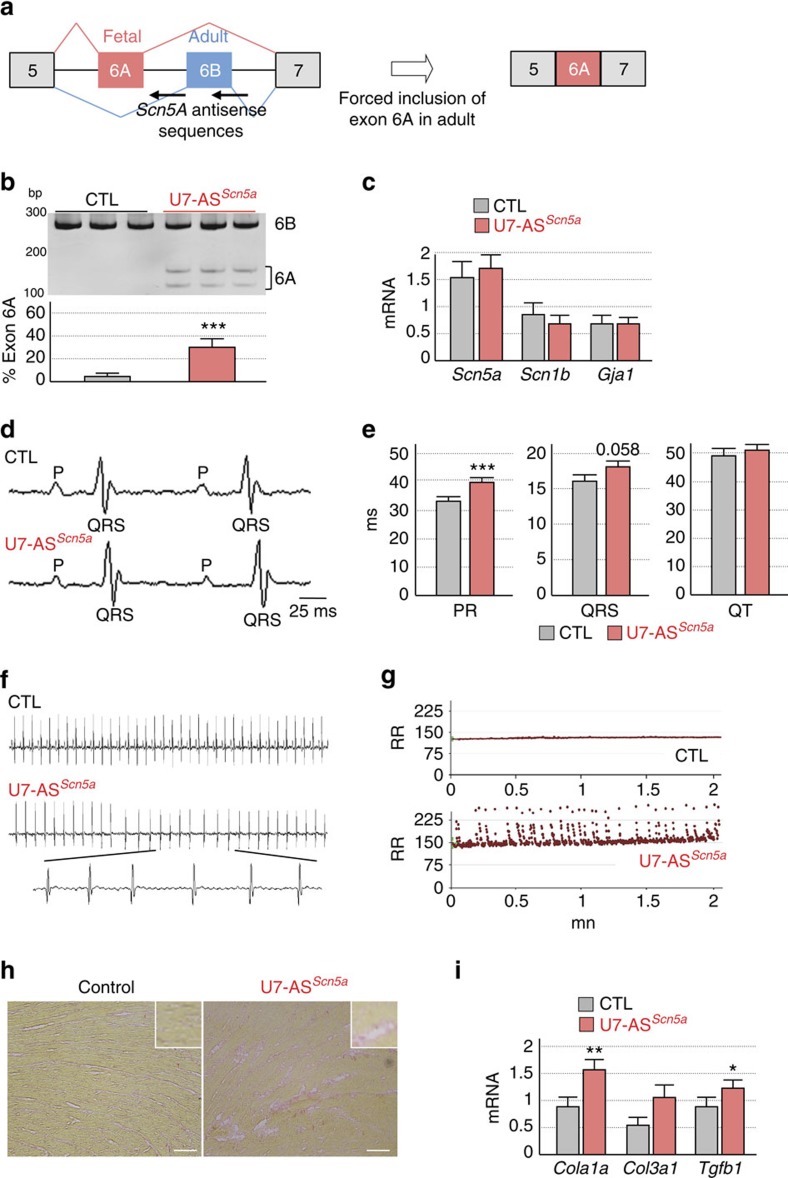
SCN5A
Analyse RNA-seq des perturbations moléculaires induites chez des patients atteints de dystrophie myotonique (2012-2016).
En collaboration avec le LBBE (Vincent Lacroix) et l'IGBMC (Nicolas Charlet-Berguerand), le PRABI-AMSB (V. Navratil, A. Campan-Fournier) a participé à l'analyse de données RNA-seq de patients atteints de Dystrophie myotonique.
Splicing misregulation of SCN5A contributes to cardiac-conduction delay and heart arrhythmia in myotonic dystrophy. Freyermuth F., Rau F., Kokunai Y., Linke T., Sellier C., Nakamori M., Kino Y, Arandel L., Jollet A., Thibault C., Philipps M., Vicaire S., Jost B., Udd B., Day J. W., Duboc D., Wahbi K., Matsumura T., Fujimura H., Mochizuki H., Deryckere F., Kimura T., Nukina N., Ishiura S., Lacroix V., Campan-Fournier A., Navratil V., Chautard E., Auboeuf D., Horie M., Imoto K., Lee K.Y, S. Swanson M., Lopez de Munain A., Inada S., Itoh H., Nakazawa K., Ashihara T., Wang E., Zimmer T., Furling D., Takahashi M.T. & Charlet-Berguerand N. Nat Commun. 2016 Apr 11;7:11067.